分子モデリング・
シミュレーションソフトウェア
Discovery Studio
創薬研究とは多目的最適化にほかなりません。研究者は生化学的有効性と、ADME や毒性といった特性の両方について最適化を行わなければなりません。Discovery Studio の最新リリースでは、業界をリードするバイオ向けシミュレーションツールに新たなサイエンスが導入された他、低分子デザインのための高度な機能にもさらなる強化が行われています。Accelrys Enterprise Platform (AEP) テクノロジーを基盤としたAccelrys DiscoveryStudio は、ライフサイエンス研究分野において最も包括的かつコラボレーション型のプラットフォームとして独自の地位を築いています
より良い生物製剤開発を迅速に実現
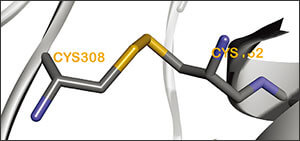
ジスルフィド架橋に適した変異サイト同定のための新技術により、分子生物学者や抗体エンジニアリング担当者は新たな生物製剤の安定性を向上させることができます。
低分子SBD 分析
最新リリースには、望ましい相互作用、望ましくない相互作用、満たされていない相互作用(相互作用する相手がいない原子)など、再設計されたデザインによる非結合相互作用モニタが用意されています。
お気軽にお問い合わせください
シミュレーションの強化
CHARMm 37b2
新しくリリースされたCHARMm では、長期間(ナノ秒)でのシミュレーションおよび系のサイズ、両面におけるパフォーマンス面での強化がなされています。
charmm36 力場をサポートします(CGenFF を含む)。
charmm36 には、強化された主鎖CMAP および側鎖二面角パラメタが含まれます。
charmm36 では、さらに、charmm22 でのタンパク質のコンフォメーションのαへリックスへのバイアスが補正されており、へリックス コイル転移における協同性の再現性が向上しています。
charmm27 力場をサポートするすべてのプロトコルはcharmm36 もサポートします。
明示的な水溶媒
パフォーマンスの向上により、完全長抗体の構造をすばやく溶媒化し、水分子と対イオンを明示的に扱ってシミュレーションを実行できます。
分子動力学ワークフロー
CHARMm およびNAMD ベースの両方のプロトコルで、分子動力学シミュレーションのデフォルト設定を強化しています。
解析を容易にするために、デフォルトで時間経過に対する温度または時間経過に対するエネルギー量がレポートに図示されるようになりました。また、フレーム毎のエネルギー値がデフォルトでCSVファイルに保存されるようになりました。
分子動力学分析
複数のDCD ファイルからフレームを選択したり、原子のサブセットを選択したりして、分析を実行できるようになりました。
実験で得られた参照構造に対するRMSD 計算、または、全フレームあるいは選択したフレーム内でのRMS 変動(RMSF) 計算が、これまで以上に使いやすくなっています。
NAMD UI の向上
外部プログラムであるNAMD9(イリノイ大学アーバナ シャンペーン校から入手できます) 実行形式との連携強化により、NAMD を簡単に利用できます。
実験で得られた参照構造に対するRMSD 計算、または、全フレームあるいは選択したフレーム内でのRMS 変動(RMSF) 計算が、これまで以上に使いやすくなっています。
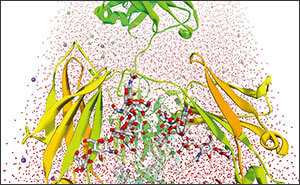
明示的な水により溶媒化された系の断面で、IgG1抗体のFc領域に結合したオリゴ糖群を示している
お気軽にお問い合わせください
バイオセラピューティックスの新技術
ジスルフィド結合予測
ジスルフィド架橋サイトの予測により、タンパク質の安定性を向上させます。
立体構造が適切な位置にある事だけでなく10 、立体的衝突、熱運動性、残基の深度11 など、その他の因子も考慮してジスルフィド結合の評価を行います。
サイトを表示、拡大して、予測した架橋を確認できます。
予測したサイトをジスフィルド架橋に変換します。
複数変異ツール
「Calculate Mutation Energy(Binding)」プロトコルおよび「Calculate Mutation Energy(Stability)」プロトコルで複数の同時変異が扱えるようになりました。
選択した残基ごとに、それぞれ異なる種類のアミノ酸を指定、変異させることができます。
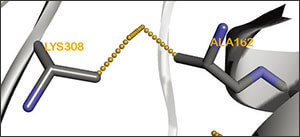
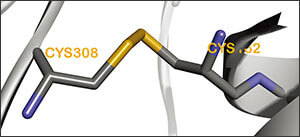
リパーゼB [PDB: 1TCA] においてトップの運動性スコアを獲得、予測されたジスフィルド架橋が、Kim らによって実際に設計された位置[12] に正確に一致
抗体モデリング ワークフロー
以下の変更があります
抗体データベースの強化により、あらかじめクリーンアップされ重ね合わされたFab およびFv テンプレート構造が提供されるようになりました。
Identify Framework Template プロトコルはヒットしたテンプレート構造をダウンロード、この構造へのリンクをレポート内に作成するため、構造へのアクセスと分析が簡単になりました。
HMMER 3.0( 隠れマルコフ モデルに基づくHMMER)が「Annotate Antibody Sequence」プロトコルおよび「Find Antibody Domains」プロトコルで使用されるようになりました。
Identify Framework Templates プロトコルによりダウンロードしたテンプレートを、フレームワーク モデルの作成に直接使用できます。
抗体モデルをインターフェース テンプレートだけで構築できるようになりました。(L 鎖 / H 鎖テンプレートの入力はオプションです)
「Model Antibody Loops」プロトコルで、入力されたタンパク質分子の残基番号を保持できます。
モデル構築プロトコルに粗視化並列処理オプションが追加されました。
お気軽にお問い合わせください
低分子SBDD強化
非結合相互作用分析
刷新された非結合相互作用モニタにより、受容体- リガンド ドッキング モデルの後処理と分析の品質が向上しています。
望ましい相互作用、望ましくない相互作用、満たされていない相互作用など、当該分野でのあらゆる最新研究を組み合わせています。
新しいAEP コンポーネントによって非結合相互作用の迅速な計算が可能になり、スクリーニング結果の後処理と絞り込みを容易に行えます。
In-situ リガンド エニュメレーション (GROW)
リガンドの優先順位付けに刷新された非結合相互作用を考慮するようになりました。
網羅的な二面角サンプリングをオプションとして実施できるようになりました。
リガンド拘束を適用して、in-situ での最適化の間、骨格の固定またはharmonic potential による抑制を行うことができます。
最適化後の骨格のRMSD の変化を計算します。
骨格ホッピング (REPLACE)
リガンドの優先順位付けに刷新された非結合相互作用を考慮するようになりました。
リガンド拘束を適用して、in-situ での最適化の間、骨格を拘束できます。
最適化後の骨格のRMSD 変化を計算します。
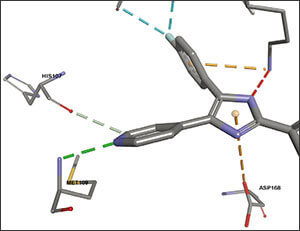
P38a と SB203580 の間に同定された非結合相互作用の例。古典的な水素結合、従来とは異なる「C-H」水素結合、および中央のイミダゾール環の結合パターンのミスマッチによる望ましくない相互作用を示す[PDB:1A9U]
リガンド ポーズの分析
望ましい相互作用、望ましくない相互作用、満たされていない相互作用など、刷新された非結合相互作用を用いる事ができるようになりました。
重要な相互作用の解析に役立つ統計情報やヒートマップを描くスクリプトも加わっています。
Matched Molecular Pair13,14,15 によるActivity Cliff の解析
結果の微調整を行うための新しいパラメタが追加されました。 Minimum Core Size、Maximum Fragment Size、およびMaximum Core Size(最大コア サイズ)です。
中核機能
非抗体データベース、Blast データベース、PharmaDB データベース更新
DS クライアントのメモリ管理の強化
クライアントのメモリ管理が大幅に強化されたため、きわめて大型の分子系を手軽に扱えるようになりました。
元に戻す(Undo)機能も改善されたため、DS クライアントのメモリ使用量は時間が経過しても大幅に増加することはありません。
DS クライアントの安定性の向上
DS4.0 では、クライアントの安定性が大幅に向上しました。
DSV ファイル形式のアップグレード
DSV ファイル形式での圧縮機能が大幅に向上したため、きわめて大規模な系も、より小さいサイズのファイルに保存できるようになりました
プラットフォーム
DS4.0 は、最新リリースのAccelrys EnterprisePlatform バージョン 9.1 を基盤として構築され、これをサポートします。
お気軽にお問い合わせください
 電話でお問い合わせ
電話でお問い合わせ
- 東京(担当:SATグループ)
- 03-3520-3082
受付時間 9:00-17:30(土・日・祝除く)

 製造業向けITソリューション
製造業向けITソリューション 品質DX支援 QX digital solution
品質DX支援 QX digital solution 建設業務改善ソリューション
建設業務改善ソリューション ビル管理業務支援 DK-CONNECT BM
ビル管理業務支援 DK-CONNECT BM FILDER SiX TOP
FILDER SiX TOP FILDER SiX 電気 TOP
FILDER SiX 電気 TOP Rebro D TOP
Rebro D TOP 実験記録をデータベース化 ParsleyLab
実験記録をデータベース化 ParsleyLab マテリアルサイエンス向けソフト Materials Studio
マテリアルサイエンス向けソフト Materials Studio 熱力学物性予測ソフトウェア COSMO
熱力学物性予測ソフトウェア COSMO 電子実験ノート
電子実験ノート 総合3DCG 制作ソフトウェア Maya
総合3DCG 制作ソフトウェア Maya 総合3DCG 制作ソフトウェア 3ds Max
総合3DCG 制作ソフトウェア 3ds Max 3Dキャラクタアニメーション制作ソフトウェア MotionBuilder
3Dキャラクタアニメーション制作ソフトウェア MotionBuilder モーションキャプチャーシステム Xsens MVN
モーションキャプチャーシステム Xsens MVN